Перейти к:
Молекулярно-генетический анализ возбудителей бактериальных пневмоний, ассоциированных с COVID-19, в стационарах г. Ростова-на-Дону
https://doi.org/2219-5238/2021-29-12-64-71
Аннотация
Введение. Присоединение бактериальной суперинфекции на фоне COVID-19 является одной из основных причин, приводящих к более тяжелому течению болезни у пациента, повышению риска неблагоприятного исхода заболевания и, как следствие, увеличению времени пребывания в стационаре. В связи с этим большое внимание исследователей уделяется изучению генетических маркеров, позволяющих выявлять клональные связи между различными изолятами возбудителей бактериальных коинфекций, что в свою очередь позволяет дифференцировать внутрибольничные и внебольничные случаи заражения.
Цель – изучение генетического разнообразия и клональных связей A. baumannii и P. aeruginosa, выделенных от пациентов с новой коронавирусной инфекцией в г. Ростове-на-Дону.
Материалы и методы. Исследовали биологический материал от 217 пациентов с диагнозом «внебольничная пневмония», находившихся на амбулаторном лечении или в стационарах г. Ростова-на-Дону. Полногеномное секвенирование штаммов A. baumannii и P. aeruginosa проведено на секвенаторе MiSeq (Illumina, США). Отбор SNP-маркеров проводили с помощью авторского программного обеспечения, написанного на языках Java и Python. Кластерный анализ и построение дендрограммы проводили с использованием авторского программного обеспечения по методу UPGMA. Для построения дендрограммы применена программа MEGA 5.
Результаты. Установлен спектр этиологических агентов бактериальной природы, вызывающих развитие вторичной инфекции на фоне COVID-19. По результатам полногеномного секвенирования 10 возбудителей пневмоний, выделенных от пациентов с новой коронавирусной инфекцией, выявлена клональность отдельных штаммов. Доказано внутрибольничное происхождение двух изолятов P. aeruginosa и двух – A. baumannii, в свою очередь анализ плазмидного состава подтвердил их внутрибольничное происхождение. Присоединение вторичной бактериальной инфекции у пациентов, находящихся на лечении, может быть обусловлено как патологическим развитием доминирующей микрофлоры слизистых верхних дыхательных путей, обеспечивающей нормобиоценоз у здоровых людей, так и несоблюдением в должном объеме принципов противоэпидемического режима и инфекционной безопасности в медицинских организациях в отношении инфекций, связанных с оказанием медицинской помощи.
Заключение. Проведенный анализ позволил определить этиологическую структуру пневмоний, у пациентов с COVID-19. Данные полногеномного секвенирования с последующим биоинформационным анализом позволили выявить внутрибольничное происхождение ряда штаммов P. aeruginosa и A. baumanii.
Ключевые слова
Для цитирования:
Носков А.К., Попова А.Ю., Водопьянов А.С., Писанов Р.В., Чемисова О.С., Павлович Н.В., Демина Ю.В., Гудуева Е.Н., Ковалев Е.В., Карпущенко Г.В. Молекулярно-генетический анализ возбудителей бактериальных пневмоний, ассоциированных с COVID-19, в стационарах г. Ростова-на-Дону. Здоровье населения и среда обитания – ЗНиСО. 2021;1(12):64-71. https://doi.org/2219-5238/2021-29-12-64-71
For citation:
Noskov A.K., Popova A.Yu., Vodop’ianov A.S., Pisanov R.V., Chemisova O.S., Pavlovich N.V., Demina Yu.V., Gudueva E.N., Kovalev E.V., Karpushchenko G.V. Molecular Genetic Analysis of the Causative Agents of COVID-19–Associated Bacterial Pneumonia in Hospitals of Rostov-on-Don. Public Health and Life Environment – PH&LE. 2021;1(12):64-71. (In Russ.) https://doi.org/2219-5238/2021-29-12-64-71
Введение. Присоединение бактериальной суперинфекции на фоне COVID-19 является одной из основных причин, приводящих к более тяжелому течению болезни у пациента, повышению риска неблагоприятного исхода заболевания и, как следствие, увеличению времени пребывания в стационаре [1–6]. Так, например, Acinetobacter baumannii, резистентный к карбопенемам, явился этиологическим фактором продолжительной вспышки внутрибольничной инфекции в Нью-Джерси, что, по мнению исследователей, было связано с его способностью длительно сохраняться на различных поверхностях и руках медицинских работников [7]. Не меньшую проблему представляет собой синегнойная палочка – Pseudomonas aeruginosa, что особенно актуально ввиду приобретения ей множественной антибиотикорезистентности. По данным ряда авторов, P. aeruginosa является одним из возбудителей бактериальных пневмоний, ассоциированных с COVID-19 [7–10].
В связи с этим большое внимание исследователей уделяется изучению генетических маркеров, позволяющих выявлять клональные связи между различными изолятами возбудителей бактериальных коинфекций, что в свою очередь позволяет дифференцировать внутрибольничные и внебольничные случаи заражения [11].
Другим важным аспектом является увеличение удельного веса патогенов, обладающих множественной лекарственной устойчивостью, обусловленной конъюгативными плазмидами [12–14]. Согласно данным литературы именно такие плазмиды являются одной из причин формирования внутрибольничных штаммов, обеспечивая их селективное преимущество перед возбудителями внебольничных инфекций [15][16]. Причем особенно остро эта проблема актуализировалась в период пандемии COVID-19 из-за существенно возросшей нагрузки на стационары. В настоящее время благодаря современным молекулярно-генетическим методам, в частности высокопроизводительному полногеномному секвенированию, становится возможным обнаружение подобных генетических элементов и раскрытие механизмов множественной антибиотикоустойчивости [17][18].
Ранее нами показано, что особенностью внебольничных пневмоний у больных с лабораторно подтвержденным COVID-19 является более высокая частота микст-инфекций как вирусной, так и бактериальной этиологии, в том числе характеризующихся полирезистентностью к антибактериальным препаратам [19].
Цель настоящего исследования состояла в изучении генетического разнообразия и клональных связей A. baumannii и P. aeruginosa, выделенных от пациентов с новой коронавирусной инфекцией в г. Ростове-на-Дону.
Материалы и методы. В период 2–24 декабря 2020 г. в г. Ростове-на-Дону выполнено исследование 217 проб мокроты от больных с внебольничной пневмонией, находившихся в двух городских больницах г. Ростова-на-Дону, обозначенных как медицинские организации «А» и «Б». Критериями включения в исследование являлись возраст старше 18 лет, установленный диагноз внебольничной пневмонии (J 18.9) согласно Российским национальным рекомендациям по внебольничной пневмонии (2019) и информированное согласие пациента на участие в исследовании. Пациентов обследовали двукратно: при поступлении в стационар и через 6–10 дней после лечения в стационаре.
Для выявления микроорганизмов на объектах окружающей среды отбирали смывы в палатах и других помещениях в соответствии с МУК 4.2.2942–11 «Методы санитарно-бактериологических исследований объектов окружающей среды, воздуха и контроля стерильности в лечебных организациях».
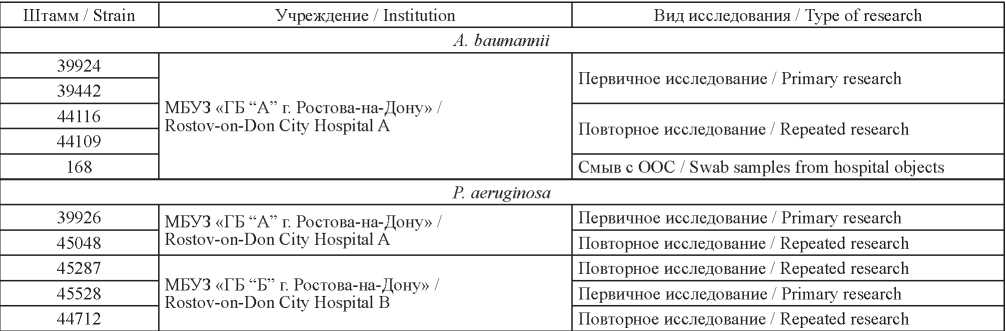
Проведен молекулярно-генетический анализ геномного разнообразия возбудителей внебольничных пневмоний, изучено пять штаммов P. aeruginosa и пять штаммов A. baumannii (табл. 1).
Таблица 1. Штаммы, использованные в работе
Table 1. Strains used in the study
Полногеномное секвенирование проведено на секвенаторе MiSeq (Illumina, США). Для тримминга использовали алгоритм Trimmomatic [20]. Сборку геномов проводили с применением геномного ассемблера Spades [21]. В сравнительный анализ включены данные полногеномного секвенирования штаммов P. aeruginosa и A. baumannii, полученные из базы данных NCBI. Отбор SNP-маркеров проводили с помощью авторского программного обеспечения, написанного на языках Java и Python [22]. Кластерный анализ и построение дендрограммы проводили с использованием авторского программного обеспечения по методу UPGMA. Для визуализации построенной дендрограммы применена программа MEGA 5 [23].
С целью амплификации плазмид из суточных культур микроорганизмов готовили взвесь 109 мк.кл./мл в жидкой среде LB, содержащей смесь антибиотиков (цефтриаксон 5 мкг/мл, хлорамфеникол 5 мкг/мл, цефотаксим 5 мкг/мл), и инкубировали при 37 °С 12 часов. После инкубации 1 мл каждой культуры переносили в пластиковые пробирки. Бактерии осаждали центрифугированием и проводили выделение ДНК с использованием набора реагентов «Рибо преп» (ООО «Амплисенс», г. Москва).
Результаты и обсуждение. В результате обследования 217 пациентов с диагнозом «внебольничная пневмония» в первый день поступления в стационар медицинских организаций «А» и «Б» наличие SARS-CoV-2 лабораторно подтверждено у 71 пациента (32,7 %). Установлено, что частота случаев ВП увеличивалась пропорционально возрасту пациентов. Так, 76,7 % случаев ВП зарегистрировано у лиц старше 50 лет, при этом наибольшее (32,5 %) число заболевших отмечено среди пациентов от 60 до 69 лет. По данным National Center for Health Statistics, у пожилых людей заболеваемость внебольничной пневмонией в два раза выше, чем у лиц молодого возраста, а сопутствующие заболевания, нередко с декомпенсацией, усложняют лечение больного и ухудшают прогноз заболевания [24]. В связи с этим особенно важно было определить спектр этиологических агентов, способных утяжелить течение внебольничной пневмонии, ассоциированной с COVID-19. В ходе проведенного бактериологического исследования мокроты от больных у 52,1 % (113 из 217) пациентов с внебольничной пневмонией были выявлены возбудители бактериальной и грибковой природы.
Следует отметить, что наиболее часто встречающийся этиологический агент внебольничных пневмоний Streptococcus pneumoniae не был обнаружен в данном исследовании, что может быть связано с высоким охватом населения Ростовской области вакцинацией от пневмококков. Наиболее частым этиологическим агентом ВП бактериальной природы являлись плазмокоагулирующие стафилококки (Staphylococcus aureus), которые обнаружены у 5,5 % пациентов. Были зарегистрированы случаи выделения представителей семейства Enterobacteriaceae, в том числе Escherichia coli (у 1,8 % пациентов) и Klebsiella pneumoniae (2,3 %), а также культур неферментирующих грамотрицательных бактерий (НГБО), преимущественно A. baumanii (2,3 %) и P. aeruginosa (1,4 %). В структуре изолированной микрофлоры превалировали грибы рода Candida, среди которых 71,4 % пришлось на долю C. albicans (рис. 1).
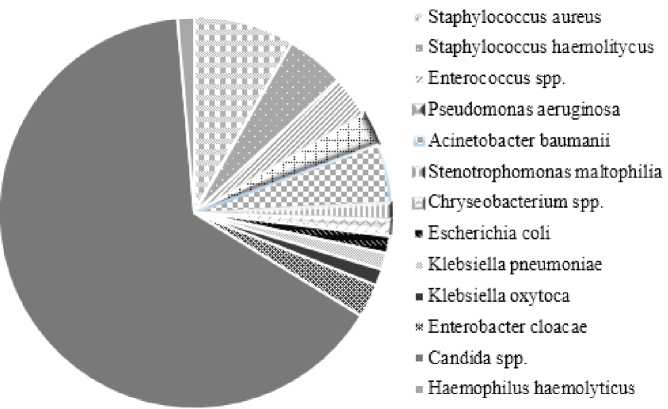
Рис. 1. Спектр этиологических возбудителей внебольничных пневмоний, ассоциированных с COVID-19
Fig. 1. Spectrum of etiological pathogens of community-acquired pneumonia associated with COVID-19
Регулярное использование инвазивных методов лечения у пациентов с SARS-CoV-2, применение в схеме лечения данной группы пациентов кортикостероидных препаратов увеличивает риск распространения внутрибольничной инфекции среди пациентов. В связи с вышеизложенным проведено повторное обследование пациентов через 6–10 дней после лечения в стационаре. В результате присоединение вторичной инфекции отмечено у 51,6 % пациентов. Из возбудителей бактериальной природы превалировали типичные представители нозокомиальных инфекций – неферментирующие грамотрицательные бактерии A. baumanii (у 8,0 % пациентов с вторичной инфекцией), P. aeruginosa (3,2 %) и K. pneumoniae (6,4 %), в том числе характеризующиеся полирезистентностью к антибактериальным препаратам.
В результате исследования смывов из ООС (палат, коридоров) изолированы устойчивые к широкому спектру антибактериальных препаратов культуры S. haemolyticus и A. baumanii.
Дальнейшая работа была направлена на анализ источника присоединения вторичной инфекции у пациентов с пневмонией, для этого отобраны штаммы A. baumanii и P. aeruginosa.
Молекулярно-генетический анализ штаммов P. aeruginosa
Проведен сравнительный анализ пяти геномов P. aeruginosa и составлен перечень SNP-маркеров для последующего анализа. По итогам этого этапа отобрано 41 898 SNP, расположенных в открытых рамках считывания.
Последующий анализ коллекции геномов P. aeruginosa, состоящей из пяти изолятов, выделенных в г. Ростове-на-Дону, и 53 штаммов из базы данных NCBI, позволил построить дендрограмму, отражающую филогенетические связи между изучаемыми штаммами (рис. 2). В ряде случаев прослеживается четкая географическая приуроченность отдельных групп штаммов. Так, например, штаммы, выделенные в Москве в 2019–2020 гг., попали в отдельный кластер, отличаясь от кластера штаммов из Нижнего Новгорода.

Рис. 2. Дендрограмма, отражающая филогенетические связи между изучаемыми штаммами P. aeruginosa
Fig. 2. Dendrogram showing phylogenetic relationships between the studied P. аeruginosa strains
Штаммы P. aeruginosa, выделенные в г. Ростовена-Дону в 2020 г., распределились между четырьмя кластерами. Изоляты P. aeruginosa №№ 44712 и 45287, выделенные в медицинской организации «Б», попали в общий кластер, отличаясь друг от друга по 13 SNP. В совокупности с данными о времени пребывания пациента в стационаре (забор мазка проводился повторно на 10-й день госпитализации) это свидетельствует о внутрибольничном заражении единым клоном P. aeruginosa, циркулирующим в данном стационаре. Необходимо отметить, что отличие описанных штаммов от изолята P. aeruginosa № 45528, выделенного в этой же больнице, но при взятии биологического материала в день поступления в стационар, составило уже более 8 тысяч SNP, что подтверждает его принадлежность к другому клону и присоединение коинфекции, по-видимому, за счет развития условно-патогенной микрофлоры на фоне поражения респираторной системы вирусом SARS-CoV-2.
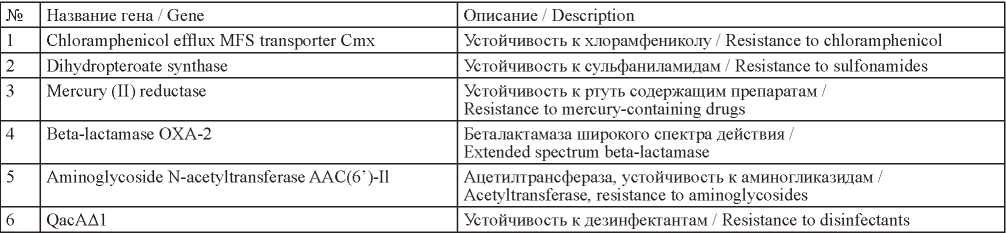
Подробный анализ полученных полногеномных последовательностей пяти штаммов P. aeruginosa позволил выявить последовательности конъюгативной плазмиды NC11MDR19 размером 19217 п.о., в которой обнаружены 35 генов, в том числе пять идентифицированы как гены множественной антибиотикоустойчивости и один ген – устойчивости к дезинфектантам (табл. 2). Весьма примечательно, что данная плазмида обнаружена в двух штаммах P. aeruginosa № 44712 и 45287, выделенных от больных, госпитализированных в медицинскую организацию «Б», что также свидетельствует в пользу их общего внутрибольничного происхождения. Следует отметить, что в смывах из объектов окружающей среды данного стационара штаммы P. aeruginosa изолированы не были. Тем не менее результаты проведенных исследований подтверждают внутрибольничное распространение возбудителя нозокомиальной инфекции P. aeruginosa. В то же время два штамма P. aeruginosa, выделенные от пациентов стационара «А», отнесены к разным клонам, т. е. доказательств внутрибольничного заражения пациента, проходящего лечение в данной медицинской организации, нами не получено.
Таблица 2. Некоторые гены плазмиды NC11MDR19
Table 2. Some genes of plasmid NC11MDR19
Молекулярно-генетический анализ штаммов A. baumannii
С целью изучения генетического разнообразия штаммов A. baumannii создана рабочая коллекция, в которую вошли пять геномов, полученных из биологического материала от больных, госпитализированных в медицинскую организацию «А», и 61 геном из базы данных NCBI. По итогам составлен перечень из 77 408 SNP, расположенных в открытых рамках считывания, что позволило построить дендрограмму, отражающую степень генетической связи между различными штаммами (рис. 3).
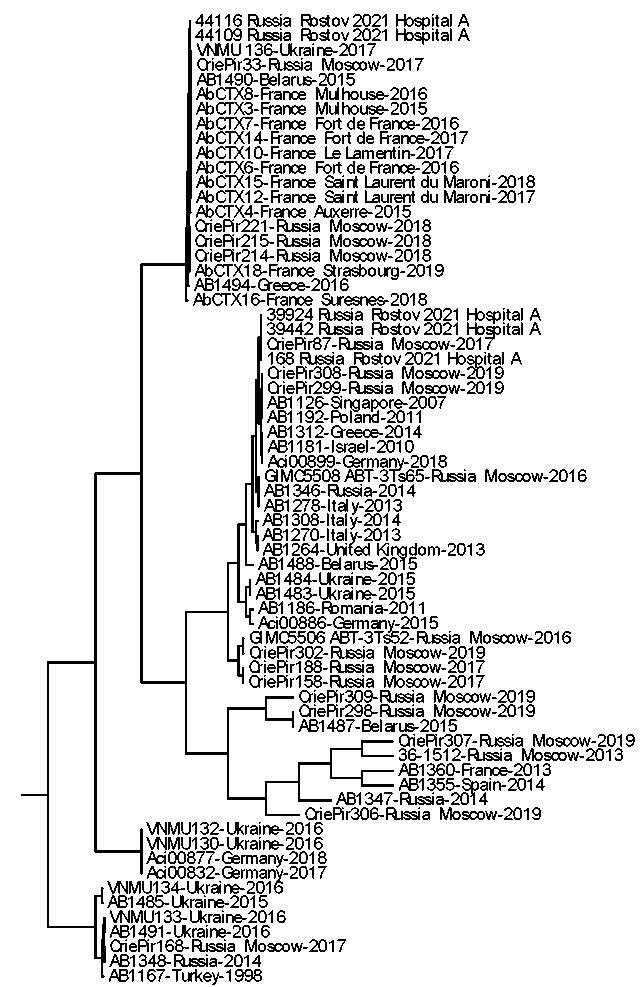
Рис. 3. Дендрограмма, отражающая филогенетические связи между изучаемыми штаммами A. baumanniii
Fig. 3. Dendrogram showing phylogenetic relationships between the studied A. baumannii strains
Анализ дендрограммы показал, что штаммы A. baumannii №№ 44116 и 44109 представляют собой один клон, отличаясь друг от друга на два SNP. При этом от наиболее генетически близкого им штамма AbCTX4, выделенного во Франции (2015 г.), их отличало уже 448 SNP. Учитывая, что данные штаммы были отобраны у пациентов на 10-й день госпитализации, это доказывает, что они представляют собой общий внутрибольничный клон, циркулирующий в медицинской организации «А».
Три штамма A. baumannii №№ 168, 39442 и 39924 образуют общий кластер, отличаясь между собой на 8-17 SNP. От штамма A. baumannii № CriePir87 их отличали 32 единичные нуклеотидные замены. Важно отметить, что два из этих штаммов – №№ 39442 и 39924 – были выделены от пациентов в момент поступления в стационар, в то время как штамм № 168 был выделен из смыва с объектов внешней среды. Сходные данные получены группой авторов при изучении штаммов A. baumannii из различных стационаров – были выявлены совпадающие PFGE-типы между клиническими изолятами и штаммами из окружающей среды [25].
Подробный биоинформационный анализ позволил выявить у двух штаммов A. baumannii №№ 44116 и 44109 конъюгативную плазмиду NC16MDR19 размером 11 194 п.о., несущую ген устойчивости к карбопенемам/пенициллинам. Учитывая, что подобные плазмиды не встречаются у природных штаммов, ее наличие в изучаемых изолятах подтверждает их внутрибольничное происхождение.
Заключение
Таким образом, в ходе проведенного исследования установлен спектр этиологических агентов бактериальной природы, вызывающих развитие вторичной инфекции на фоне COVID-19. По результатам полногеномного секвенирования 10 возбудителей пневмоний, выделенных от пациентов с новой коронавирусной инфекцией, выявлена клональность отдельных штаммов. Доказано внутрибольничное происхождение двух изолятов P. aeruginosa и двух – A. baumanii, в свою очередь анализ плазмидного состава подтвердил их внутрибольничное происхождение.
Присоединение вторичной бактериальной инфекции у пациентов, находящихся на лечении, может быть обусловлено разными сценариями, одним из которых является патологическое развитие доминирующей микрофлоры слизистых верхних дыхательных путей, обеспечивающей нормобиоценоз у здоровых людей, чему способствует назначение пациентам стероидов и ингибиторов провоспалительных цитокинов, а также эмпирическое применение антибактериальных препаратов широкого спектра действия у пациентов с внебольничными пневмониями, в том числе ассоциированными с COVID-19. В то же время несоблюдение в должном объеме принципов противоэпидемического режима и инфекционной безопасности в медицинских организациях в отношении инфекций, связанных с оказанием медицинской помощи (ИСМП), также может служить причиной развития внутрибольничных инфекций у пациентов. Очевидно, что данная ситуация не уникальна, ИСМП являются одной из ведущих проблем здравоохранения. В свою очередь всестороннее изучение и обсуждение факторов регистрации ИСМП, условий, приводящих к их возникновению и распространению в медицинских организациях, в конечном итоге способствуют совершенствованию мероприятий в рамках обеспечения инфекционной безопасности в стационарах.
Список литературы
1. Durán-Manuel EM, Cruz-Cruz C, Ibáñez-Cervantes G, et al. Clonal dispersion of Acinetobacter baumannii in an intensive care unit designed to patients COVID-19. J Infect Dev Ctries. 2021;15(1):58–68. doi: 10.3855/jidc.13545
2. Mirzaei R, Goodarzi P, Asadi M, et al. Bacterial co-infections with SARS-CoV-2. IUBMB Life. 2020;72(10):2097–2111. doi: 10.1002/iub.2356
3. Nasr P. Genetics, epidemiology, and clinical manifestations of multidrug-resistant Acinetobacter baumannii. J Hosp Infect. 2020;104(1):4–11. doi: 10.1016/j.jhin.2019.09.021
4. Mohd Sazlly Lim S, Zainal Abidin A, Liew SM, Roberts JA, Sime FB. The global prevalence of multidrug-resistance among Acinetobacter baumannii causing hospital-acquired and ventilator-associated pneumonia and its associated mortality: A systematic review and meta-analysis. J Infect. 2019;79(6):593–600. doi: 10.1016/j.jinf.2019.09.012
5. Эсауленко Н.Б., Ткаченко О.В. Изменение чувствительности бактериальной флоры у больных Covid-19. Результаты собственных исследований // Клиническая лабораторная диагностика. 2021. Т. 66. № S4. C. 80.
6. Бисенова Н.М., Ергалиева А.С. Микробиологические показатели пациентов с подтвержденной инфекцией Covid-19 // Наука и Здравоохранение. 2020. № 6. С. 5–10.
7. Perez S, Innes GK, Walters MS. et al. Increase in hospital-acquired carbapenem-resistant Acinetobacter baumannii Infection and colonization in an acute care hospital during a surge in COVID-19 admissions – New Jersey, February–July 2020. MMWR Morb Mortal Wkly Rep. 2020;69(48):1827–1831. doi: 10.15585/mmwr.mm6948e1
8. Baiou A, Elbuzidi AA, Bakdach D, et al. Clinical characteristics and risk factors for the isolation of multi-drug-resistant Gram-negative bacteria from critically ill patients with COVID-19. J Hosp Infect. 2021;110:165–171. doi: 10.1016/j.jhin.2021.01.027
9. Wieland K, Chhatwal P, Vonberg RP. Nosocomial outbreaks caused by Acinetobacter baumannii and Pseudomonas aeruginosa: Results of a systematic review. Am J Infect Control. 2018;46(6):643–648. doi: 10.1016/j.ajic.2017.12.014
10. Ronda VE, Alcaraz SR, Torregrosa PR, et al. Application of validated severity scores for pneumonia caused by SARS-CoV-2. Med Clin (Barc). 2021;157(3):99–105. doi: 10.1016/j.medcli.2021.01.002
11. Acosta F, Fernández-Cruz A, Maus SR, et al. In-depth study of a nosocomial outbreak caused by extensively drug-resistant Pseudomonas aeruginosa using whole genome sequencing coupled with a polymerase chain reaction targeting strain-specific single nucleotide polymorphisms. Am J Epidemiol. 2020;189(8):841–849. doi: 10.1093/aje/kwaa025
12. Makke G, Bitar I, Salloum T, et al. Whole-genome-sequence-based characterization of extensively drug-resistant Acinetobacter baumannii hospital outbreak. mSphere. 2020;5(1):e00934–19. doi: 10.1128/mSphere.00934-19
13. Hujer AM, Higgins PG, Rudin SD, et al. Nosocomial outbreak of extensively drug-resistant Acinetobacter baumannii isolates containing blaOXA-237 carried on a plasmid. Antimicrob Agents Chemother. 2017;61(11):e00797–17. doi: 10.1128/AAC.00797-17
14. Botelho J, Grosso F, Peixe L. Antibiotic resistance in Pseudomonas aeruginosa – Mechanisms, epidemiology and evolution. Drug Resist Updat. 2019;44:100640. doi: 10.1016/j.drup.2019.07.002
15. Loraine J, Heinz E, Soontarach R, et al. Genomic and phenotypic analyses of Acinetobacter baumannii isolates from three tertiary care hospitals in Thailand. Front Microbiol. 2020;11:548. doi: 10.3389/fmicb.2020.00548
16. Khatun MN, Farzana R, Lopes BS, Shamsuzzaman SM. Molecular characterization and resistance profile of nosocomial Acinetobacter baumannii intensive care unit of tertiary care hospital in Bangladesh. Bangladesh Med Res Counc Bull. 2015;41(2):101–107. doi: 10.3329/bmrcb.v41i2.29991
17. Murata K, Inoue Y, Kaiho M, et al. Genomic analysis of antibiotic resistance for Acinetobacter baumannii in a critical care center. Acute Med Surg. 2019;7(1):e445. doi: 10.1002/ams2.445
18. Wareth G, Brandt C, Sprague LD, Neubauer H, Pletz MW. Spatio-temporal distribution of Acinetobacter baumannii in Germany – A comprehensive systematic review of studies on resistance development in humans (2000– 2018). Microorganisms. 2020;8(3):375. doi: 10.3390/microorganisms8030375
19. Попова А.Ю., Ежлова Е.Б., Демина Ю.В. и др. Особенности этиологии внебольничных пневмоний, ассоциированных с COVID-19. Проблемы особо опасных инфекций. 2020. № 4. С. 99–105. doi: 10.21055/0370-1069-2020-4-99-105
20. Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014;30(15):2114–2120. doi: 10.1093/bioinformatics/btu170
21. Bankevich A, Nurk S, Antipov D, et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. J Comput Biol. 2012;19(5):455–477. doi: 10.1089/cmb.2012.0021
22. Водопьянов А.С., Писанов Р.В., Водопьянов С.О., Олейников И.П. Совершенствование методики SNP-типирования штаммов Vibrio cholerae на основе анализа первичных данных полногеномного секвенирования // Журнал микробиологии, эпидемиологии и иммунобиологии. 2020. Т. 97. № 6. С. 587–593. doi: 10.36233/0372-9311-2020-97-6-9
23. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol. 2011;28(10):2731–2739. doi: 10.1093/molbev/msr121
24. Яковлев С.В. Внебольничная пневмония у пожилых: особенности этиологии, клинического течения и антибактериальной терапии // Русский Медицинский Журнал № 16 от 16.08.1999. С. 763.
25. Boral B, Unaldi Ö, Ergin A, Durmaz R, Eser ÖK; Acinetobacter Study Group [Corporate Author]. A prospective multicenter study on the evaluation of antimicrobial resistance and molecular epidemiology of multidrug-resistant Acinetobacter baumannii infections in intensive care units with clinical and environmental features. Ann Clin Microbiol Antimicrob. 2019;18(1):19. doi: 10.1186/s12941-019-0319-8
Об авторах
А. К. НосковРоссия
Носков Алексей Кимович – кандидат медицинских наук, директор
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002
А. Ю. Попова
Россия
Попова Анна Юрьевна – доктор медицинских наук, профессор Российская медицинская академия непрерывного профессионального образования
ул. Баррикадная, д. 2/1, г. Москва, 125993
А. С. Водопьянов
Россия
Водопьянов Алексей Сергеевич – кандидат медицинских наук, старший научный сотрудник лаборатории молекулярной биологии природно-очаговых и зоонозных инфекций
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002
Р. В. Писанов
Россия
Писанов Руслан Вячеславович – кандидат медицинских наук, заведующий лаборатории молекулярной биологии природно-очаговых и зоонозных инфекций
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002
О. С. Чемисова
Россия
Чемисова Ольга Сергеевна – кандидат биологических наук, заведующая лабораторией «Коллекция патогенных микроорганизмов»
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002
Н. В. Павлович
Россия
Павлович Наталья Владимировна – доктор медицинских наук, заведующая лабораторией
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002
Ю. В. Демина
Россия
Демина Юлия Викторовна – доктор медицинских наук, профессор
ул. Баррикадная, д. 2/1, г. Москва, 125993
Е. Н. Гудуева
Россия
Гудуева Елена Николаевна – младший научный сотрудник «Коллекция патогенных микроорганизмов»
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002
Е. В. Ковалев
Россия
Ковалев Евгений Владимирович – руководитель
ул. 18-я линия, д. 17, г. Ростов-на-Дону, 344019
Г. В. Карпущенко
Россия
Карпущенко Гарри Викторович – кандидат медицинских наук, главный врач
ул. 7-я линия, д. 67, г. Ростов-на-Дону, 344019
Рецензия
Для цитирования:
Носков А.К., Попова А.Ю., Водопьянов А.С., Писанов Р.В., Чемисова О.С., Павлович Н.В., Демина Ю.В., Гудуева Е.Н., Ковалев Е.В., Карпущенко Г.В. Молекулярно-генетический анализ возбудителей бактериальных пневмоний, ассоциированных с COVID-19, в стационарах г. Ростова-на-Дону. Здоровье населения и среда обитания – ЗНиСО. 2021;1(12):64-71. https://doi.org/2219-5238/2021-29-12-64-71
For citation:
Noskov A.K., Popova A.Yu., Vodop’ianov A.S., Pisanov R.V., Chemisova O.S., Pavlovich N.V., Demina Yu.V., Gudueva E.N., Kovalev E.V., Karpushchenko G.V. Molecular Genetic Analysis of the Causative Agents of COVID-19–Associated Bacterial Pneumonia in Hospitals of Rostov-on-Don. Public Health and Life Environment – PH&LE. 2021;1(12):64-71. (In Russ.) https://doi.org/2219-5238/2021-29-12-64-71









.png)



























