Перейти к:
Оценка генетического разнообразия возбудителя норовирусной инфекции в пунктах временного размещения беженцев в Ростовской области в 2022 году с помощью онлайн-программы NoroNetRus
https://doi.org/10.35627/2219-5238/2022-30-11-82-88
Аннотация
Введение. Очевидно, что эффективное расследование вспышек норовирусной инфекции невозможно без методов, способных дифференцировать генотипы возбудителя, основным из которых является определение типа капсида и полимеразы на основе данных секвенирования. Однако в последнее время наблюдается неустойчивая работа международных сервисов по оценке геномов норовируса с ряда IP-адресов, находящихся в Российской Федерации, что может оказать влияние на оперативность расследования вспышек норовирусной инфекции в нашей стране.
Цель – разработка отечественного программного обеспечения для генотипирования норовирусов и оценка с его помощью генетического разнообразия возбудителя норовирусной инфекции, выявленного в 2022 году в Ростовской области.
Материалы и методы. Материалом для исследования послужили образцы фекалий от 210 больных с симптомами острых кишечных инфекций из пунктов временного размещения беженцев и детских организованных коллективов. Контингент обследуемых – пациенты в возрасте от 1 месяца до 74 лет с симптомами острых кишечных инфекций в 2022 г. Выявление РНК возбудителя норовирусной инфекции проводили с использованием набора реагентов «АмплиСенс® ОКИ скрин-FL» (Москва, Россия). Фрагментарное секвенирование проводили на геномном анализаторе SeqStudio (Thermo Fisher, США). Авторское программное обеспечение разрабатывали на языках программирования Java и Python.
Результаты. Создана отечественная программа NoroNetRus, позволяющая определять тип капсида и полимеразы норовируса исходя из данных секвенирования. Программа бесплатна и доступна в онлайн-формате по адресу: http://antiplague.ru/noronetrus. Проведенное фрагментарное секвенирование последовательностей гена VP1 для 25 образцов норовируса показало, что они принадлежали трем разным генотипам (GII.4 Sydney, GII.10 и GII.17), причем генотип GII.10, в свою очередь, распределился между двумя разными кластерами. Полученные данные свидетельствуют о существовании как минимум четырех различных источников инфицирования.
Заключение. Полученные данные свидетельствуют о циркуляции в 2022 году в Ростовской области как минимум четырех различных геновариантов возбудителя норовирусной инфекции.
Для цитирования:
Водопьянов А.С., Писанов Р.В., Водопьянов С.О., Чемисова О.С., Герасименко А.А., Носков А.К., Слись С.С., Ненадская С.А., Коренева А.Д., Коломойцева А.В., Ковалев Е.В., Литовко А.Р., Половинка Н.В. Оценка генетического разнообразия возбудителя норовирусной инфекции в пунктах временного размещения беженцев в Ростовской области в 2022 году с помощью онлайн-программы NoroNetRus. Здоровье населения и среда обитания – ЗНиСО. 2022;1(11):82-88. https://doi.org/10.35627/2219-5238/2022-30-11-82-88
For citation:
Vodop’ianov A.S., Pisanov R.V., Vodop’ianov S.O., Chemisova O.S., Gerasimenko A.A., Noskov A.K., Slis S.S., Nenadskaya S.A., Koreneva A.D., Kolomoitseva A.V., Kovalev E.V., Litovko A.R., Polovinka N.V. Assessment of Genetic Diversity of Noroviruses Circulating in Temporary Accommodation Centers for Refugees in the Rostov Region in 2022 Using the NoroNetRus Online Software. Public Health and Life Environment – PH&LE. 2022;1(11):82-88. (In Russ.) https://doi.org/10.35627/2219-5238/2022-30-11-82-88
Введение. Норовирус является одной из ведущих причин острого небактериального эпидемического гастроэнтерита во всем мире, поскольку имеет несколько путей передачи и высокую устойчивость к дезинфицирующим средствам [1][2]. Ежегодно в мире фиксируется миллиард случаев этой инфекции, среди которых 200 000 со смертельным исходом1 . В 2020 г. в Российской Федерации зарегистрировано 103 очага групповой заболеваемости НВИ с общим числом пострадавших 1480 человек (в 2019 г. – 215 и 2705 соответственно)2 .
Немаловажно, что смена или появление нового генотипа норовируса приводит к росту как спорадической, так и групповой заболеваемости вплоть до возникновения новых эпидемических вариантов вируса, склонных к быстрому распространению [3–5]. Норовирусы представляют собой группу генетически разнообразных, одноцепочечных РНК-вирусов. Род Norovirus относится к семейству Caliciviridae и содержит только один вид: вирус Norwalk.
В 2013 г. международная рабочая группа по классификации норовирусов предложила универсальную номенклатуру обозначения генотипа, включающую определение типа капсида (G-тип) и полимеразы (P-тип) [6]. Согласно современной классификации норовирусы подразделяются на десять геногрупп (GI-GVI) и 48 генотипов. Из этих семи геногрупп только GI, GII и GIV содержат человеческие норовирусы, причем вторая геногруппа (GII) встречается в 10 раз чаще [7]. Существует восемь генотипов капсида, принадлежащих GI, и 21, принадлежащий GII. С 2001 г. большинство вспышек во всем мире вызвано вирусами GII.4, хотя периодически появляются другие генотипы, такие как GII.17 и GII.2 [1].
Очевидно, что эффективное расследование вспышек норовирусной инфекции невозможно без методов, способных дифференцировать генотипы возбудителя, что может быть полезным при выявлении связей между отдельными очагами инфекции и путей передачи возбудителя.
Одним из наиболее широко распространенных подходов для оценки получаемых данных является использование онлайн программы Norovirus Genotyping tool, позволяющей быстро определять G и P тип вируса, исходя из данных секвенирования. Программа рассчитана на работу в режиме онлайн и расположена на сервере Национального института общественного здравоохранения и окружающей среды (Нидерланды) (http://www.rivm.nl/mpf/norovirus/typingtool). Аналогичный сервис представлен на сайте Centers for Disease Control and Prevention (США) (https://norovirus.ng.philab.cdc.gov/bctyping.html). Однако в последнее время наблюдается неустойчивая работа указанных сервисов и их периодическая недоступность с ряда IP-адресов, находящихся в Российской Федерации, что может оказать влияние на оперативность расследования вспышек НВИ в нашей стране. Помимо этого, одной из целей Указа Президента от 7 мая 2018 г. № 2043 «О национальных целях и стратегических задачах…» является использование преимущественно отечественного программного обеспечения государственными органами, органами местного самоуправления и организациями.
В связи с этим цель настоящего исследования состояла в разработке отечественного программного обеспечения для генотипирования норовирусов и оценке с его помощью генетического разнообразия возбудителя норовирусной инфекции, выявленного в 2022 г. в Ростовской области.
Материалы и методы. Контингент обследуемых – пациенты в возрасте от 1 месяца до 74 лет с симптомами острых кишечных инфекций (ОКИ) в 2022 г. Материалом для исследования послужили образцы фекалий от 210 больных с симптомами ОКИ из пунктов временного размещения беженцев и детских организованных коллективов. Все пациенты и их законные представители подписали добровольное информированное согласие на участие в исследовании. Выявление РНК возбудителя норовирусной инфекции проводили с использованием набора реагентов «АмплиСенс® ОКИ скрин-FL» (Москва, Россия). Фрагментарное секвенирование проводили на геномном анализаторе SeqStudio (Thermo Fisher, США) с использованием праймеров, предложенных Cannon JL (2017).
Геномные последовательности возбудителя норовирусной инфекции для определения генотипов и создания собственного программного обеспечения получали из базы данных NCBI. Для сравнительного анализа геномов норовируса использовали программу Mega [8]. Для построения дендрограммы применяли алгоритм UPGMA. Авторское программное обеспечение разрабатывали на языках программирования Java и Python. Для выравнивания нуклеотидных последовательностей в рамках создаваемого программного обеспечения использовали модуль Scikit [9].
Результаты. Первый этап работы состоял в составлении локальной базы данных референсных последовательностей. С этой целью былииспользованы обновленные данные по классификации норовирусов, предложенные Chhabra P и соавт. [10]. Для этого из базы данных NCBI были получены геномы норовируса каждого G и P типа, и после их выравнивания получены последовательности генов VP1 и RdPr.
Согласно предлагаемому нами алгоритму, каждая анализируемая последовательность норовируса поочередно выравнивается с помощью локального выравнивания по алгоритму Смитта – Ватермана с аффинными штрафами за вставки и делеции с каждой последовательностью из созданной локальной базы данных. В качестве критерия оценки используется количество совпавших нуклеотидов, что позволяет определить наиболее близкородственную референсную последовательность. Соответственно, поочередное проведение выравнивания с последовательностями генов капсида (VP1) и полимеразы (RdPr) позволяет определить G и P тип анализируемого норовируса.
С этой целью на языке программирования Python нами разработана программа NoroNetRus, предназначенная для работы в онлайн-формате, и расположена на сайте института по адресу: http://antiplague.ru/noronetrus. Внешний вид программы представлен на рис. 1.

Рис. 1. Внешний вид стартового окна программы NoroNetRus
Fig. 1. The start window of the NoroNetRus software
Программа имеет интуитивно-понятный интерфейс, предназначена для работы через любой браузер с поддержкой JavaScript. В качестве входных данных используется fasta-файл, содержащий данные фрагментарного секвенирования норовируса. Время на анализ одной последовательности составляет от 0,5 до 3 секунд. Итогом работы программы являются типы генов VP1 и RdPr, например «Vp1: GII.17, RdRp: GII.P3».
Для валидации работы программы из базы данных NCBI нами были скачаны 100 случайных геномов норовируса и проведена виртуальная ПЦР in silico с праймерами, захватывающими фрагмент обоих генов [11]. Полученные ампликоны (фрагменты ДНК, ограниченные праймерами) были поочередно проверены в программах NoroNet (Нидерланды) и разработанной нами NoroNetRus.
По итогам валидации программа NoroNetRus на первом этапе определила G и P тип для всех последовательностей, в то время как алгоритм NoroNet (Нидерланды) не определил тип вируса для 8 последовательностей. В оставшихся 92 последовательностях обнаружено всего одно несоответствие – тип полимеразы в геноме вируса, депонированном в NCBI под номером KY424345, разработанная нами программа определила как GII.P7, в то время как программа NoroNet определила тип как GII.P6. Важным преимуществом является невозможность определения субтипов варианта капсида GII.4 при использовании программы NoroNet, что ограничивает возможности валидации. В то же время разработанная нами программа NoroNetRus успешно решает эту задачу.
При обследовании 210 пациентов с симптомами ОКИ в марте 2022 г. нами было выявлено 110 положительных проб, содержащих РНК норовируса в пяти эпидемических очагах. Два очага сформировались в пунктах временного размещения беженцев (ПВР-1 и ПВР-2), прибывших на территорию Ростовской области из Донецкой и Луганской народных республик. В этот же период было выявлено еще три очага в детских образовательных учреждениях (ДОУ) г. Ростова-на-Дону и г. Батайска. Для оценки генетического разнообразия нами проведено фрагментарное секвенирование участка гена VP1 (основной белок капсида) у 25 изолятов вируса.
Использование разработанной нами программы NoroNetRus позволило установить тип капсида для всех 25 образцов (таблица).
Таблица. Генотипы изученных изолятов норовируса
Table. Genotypes of the norovirus isolates studied
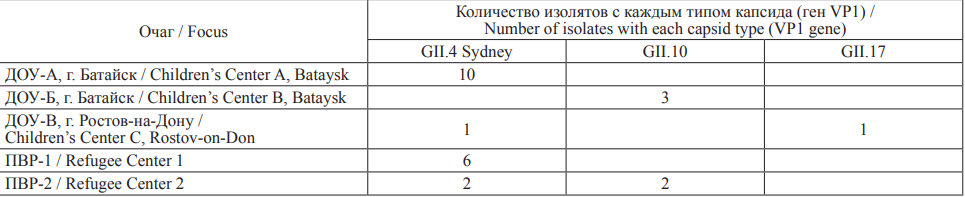
Обращает на себя внимание, что в ряде очагов циркулировали изоляты вируса, принадлежащие к одному генотипу: так, в ДОУ-А и ПВР-1 выявлена циркуляция генотипа GII.4 Sydney, а в ДОУ-Б – генотипа GII.10. В то же время в двух других очагах наблюдалась одновременная циркуляция сразу нескольких генотипов: в ДОУ-В выявлены генотипы GII.4 Sydney и GII.17, а в ПВР-2 – GII.4 Sydney и GII.10.
Для анализа мирового распространения норовируса из базы данных NCBI были получены 5947 последовательностей гена VP1 различных штаммов и с помощью разработанного нами программного обеспечения определен их генотип (рис. 2). Установлено, что наиболее распространенным в мире явился генотип GII.4 Sydney, к которому отнесено 17 % всех последовательностей. В России доминирующим явился генотип GII.3 (22 %), в то время как GII.4 Sydney занимает 4-е место по частоте встречаемости (12 %). За последние 5 лет выделение штаммов данного генотипа в России зарегистрировано в Новосибирске и Нижнем Новгороде.
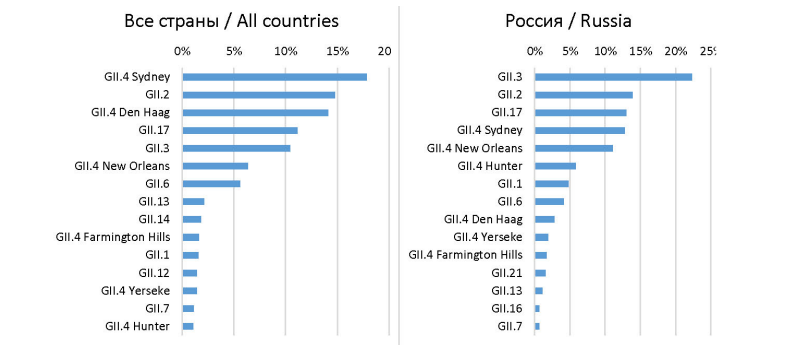
Рис. 2. Частота обнаружения различных генотипов в мире и в России (указаны первые 15 самых частых генотипов)
Fig. 2. Frequency of detection of 15 most frequent norovirus genotypes in the world and in Russia
Частота встречаемости генотипа GII.10 составила 0,02 % для мировой коллекции и 0,04 % – для России. За последние годы выделение штаммов GII.10 зарегистрировано в Испании (2019–2020 гг.) и Эфиопии (2016 г.). В России данный вариант выделялся в Челябинске в 2005 г.
Обсуждение. Норовирусы генотипа GII.17, по данным литературы, явились причиной вспышек в Хабаровском крае в 2015 г. и Амурской области в 2018 г. [12]. Интересно отметить, что норовирус GII.17 стал доминирующим генотипом в Осаке (Япония) [13], Южной Корее [14], Канаде [15], Италии [16], заменив генотип GII.4.
Вместе с тем на ряде территорий регистрируется одновременная циркуляция большого числа генотипов норовируса. Так, в Ботсване в период 2013–2015 гг., несмотря на доминирование генотипа GII.4, наблюдалось выделение минорных генотипов GII.2, GII.12, GI.9, GII.6, GII.10 [17]. Сходная ситуация наблюдалась в Эфиопии [18], Корее [19], ЮАР [20], Канаде [21].
Интересно, что отдельные вспышки, как правило, вызываются одним типом норовируса. Так, например, водная вспышка в г. Хабаровске в 2019 году была вызвана генотипом GII.P7-GII.6 [22]. Вспышка НВИ, ассоциированная с употреблением мороженного, инфекции в Бразилии в 2020 году была вызвана генотипом GII.12 [23]. Это позволяет использовать генотипирование в качестве удобного инструмента для выявления источника заражения, что и было с успехом продемонстрировано в Китае в 2017 году – проведенный анализ показал, что развитие вспышки норовируса GII.P17-GII.17 было связано с системой водоснабжения [24].
Для более точного анализа проведено выравнивание полученных фрагментов гена VP1 и построена дендрограмма, отражающая степень генетической близости между изучаемыми изолятами (рис. 3). К сожалению, в базе данных NCBI отсутствуют последовательности вируса, выделенные в Южном федеральном округе России и на Украине, что ограничивает возможности проведения анализа. При добавлении в перечень последовательностей штаммов из сопредельных государств наиболее генетически близкородственными оказались последовательности вируса, выделенного в Республике Беларусь в 2021 г. (данные не представлены).

Рис. 3. Дендрограмма, построенная на основе выравнивания фрагментов гена VP1. Для каждого изолята
указаны номер, место выделения и генотип (алгоритм Neighbor Joining)
Примечание: PVR – пункт временного размещения беженцев; DOU – детский центр.
Fig. 3. Dendrogram based on alignment of VP1 gene fragments. For each isolate, the number, place of isolation,
and genotype are shown (Neighbor Joining algorithm)
Notes: PVR – a temporary accommodation center for refugees; DOU – a children’s center.
Обращает на себя внимание, что по данным дендрограммы штаммы норовируса генотипа GII.10 образуют два разных кластера: в один кластер попали варианты вируса из ДОУ-Б, в другой – из ПВР-2. При проведении выравнивания нуклеотидных последовательностей 4774 (ПВР-2) и Е1604 (ДОУ-Б) выявлено сходство 89,7 %, при этом между собой последовательности 4774 и 4779 совпадали на 100 %. Примечательно, что поиск с использованием алгоритма BLAST не нашел близкородственных штаммов для вирусов из ПВР-2, что может свидетельствовать о новом подтипе GII.10, не включенном в существующую международную классификацию.
Аналогичная гетерогенность внутри одного генотипа была показана ранее на примере норовируса GII.17. Так, было показано, что данный генотип можно разделить на четыре различные клады [25]. В последующем было установлено, что скорость эволюции гена VP1 норовируса генотипа составляет 1,14 × 10–3 нуклеотидных замен/год [26].
Исследованные нами варианты норовируса GII.4 Sydney образовали единую группу, имея не более 0,5 % отличий друг от друга.
Заключение. Таким образом, в ходе проведенной работы был создан набор референсных последовательностей генов VP1 и RdPr норовируса второй геногруппы. Разработан алгоритм анализа данных секвенирования возбудителя норовирусной инфекции и создана отечественная программа NoroNetRus, позволяющая определять тип капсида и полимеразы норовируса, исходя из данных секвенирования. Программа бесплатнаи доступна в онлайн-формате по адресу: http://antiplague.ru/noronetrus.
Проведена валидация работы программы с использованием 100 случайных последовательностей из базы данных NCBI, подтвердившая корректность проводимого анализа.
Было проведено фрагментарное секвенирование последовательностей гена VP1 для 25 образцов норовируса, выделенных в марте 2022 г. из пунктов временного размещения беженцев и детских образовательных учреждений (ДОУ) г. Ростова-на-Дону и г. Батайска. Показано, что они принадлежали к трем разным генотипам (GII.4 Sydney, GII.10 и GII.17), причем генотип GII.10, в свою очередь, распределился между двумя разными кластерами. Полученные данные свидетельствуют о существовании как минимум четырех различных источников инфицирования
1. МУ 3.1.1.2969—11 «Эпидемиологический надзор, лабораторная диагностика и профилактика норовирусной инфекции»: Методические указания. М.: Федеральный центр гигиены и эпидемиологии Роспотребнадзора, 2012. 36 с.
2. Государственный доклад «О состоянии санитарно-эпидемиологического благополучия населения в Российской Федерации в 2020 году». [Электронный ресурс]. Режим доступа: https://www.rospotrebnadzor.ru/documents/details. php?ELEMENT_ID=18266 (дата обращения: 10.04.2022). 3 Указ Президента РФ от 7 мая 2018 г. № 204 «О национальных целях и стратегических задачах развития Российской Федерации на период до 2024 года» (с изменениями и дополнениями). [Электронный ресурс]. Режим доступа: https:// base.garant.ru/71937200/ (дата обращения: 10.04.2022).
Список литературы
1. Pires SM, Fischer-Walker CL, Lanata CF, et al. Aetiology-specific estimates of the global and regional incidence and mortality of diarrhoeal diseases commonly transmitted through food. PLoS One. 2015;10(12):e0142927. doi: 10.1371/journal.pone.0142927
2. Бутакова Л.В., Сапега Е.Ю., Троценко О.Е. и др. Генотипы норовирусов, обусловившие заболеваемость острыми кишечными инфекциями в Хабаровском крае // Здоровье населения и среда обитания. 2018. № 7. С. 52–56.
3. Medici MC, Tummolo F, De Grazia S, et al. Epidemiological dynamics of norovirus GII.4 variant New Orleans 2009. J Gen Virol. 2015;96(9):2919-2927. doi: 10.1099/vir.0.000204
4. Chong PP, Atmar RL. Norovirus in health care and implications for the immunocompromised host. Curr Opin Infect Dis. 2019;32(4):348-355. doi: 10.1097/QCO.0000000000000557
5. Kwok K, Niendorf S, Lee N, et al. Increased detection of emergent recombinant norovirus GII.P16-GII.2 strains in young adults, Hong Kong, China, 2016–2017. Emerg Infect Dis. 2017;23(11):1852-1855. doi: 10.3201/eid2311.170561
6. Сапега Е.Ю., Бутакова Л.В., Троценко О.Е. и др. Эпидемиологические аспекты норовирусной инфекции на современном этапе // Дальневосточный журнал инфекционной патологии. 2021. Т. 40. № 40. С. 72–78.
7. Хохлова Н.И., Капустин Д.В., Краснова Е.И., Извекова И.Я. Норовирусная инфекция (обзор литературы) // Журнал инфектологии. 2018. Т. 10. № 1. С. 5–14. doi: 10.22625/2072-6732-2018-10-1-5-14
8. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol. 2011;28(10):2731-2739. doi: 10.1093/molbev/msr121
9. Pedregosa F, Varoquaux G, Gramfort A, et al. Scikit-learn: machine learning in Python. J Mach Learn Res. 2011;12(85):2825-2830.
10. Chhabra P, de Graaf M, Parra GI, et al. Updated classification of norovirus genogroups and genotypes. J Gen Virol. 2019;100(10):1393-1406. doi: 10.1099/jgv.0.001318
11. Cannon JL, Barclay L, Collins NR, et al. Genetic and epidemiologic trends of norovirus outbreaks in the United States from 2013 to 2016 demonstrated emergence of novel GII.4 recombinant viruses. J Clin Microbiol. 2017;55(7):2208-2221. doi: 10.1128/JCM.00455-17
12. Сапега Е.Ю., Бутакова Л.В., Троценко О.Е., Зайцева Т.А., Курганова О.П., Копылов П.В. Заболеваемость острыми кишечными инфекциями, вызванными вирусными возбудителями, в субъектах Дальневосточного федерального округа // Дальневосточный журнал инфекционной патологии. 2021. Т. 41. № 41. С. 36–43.
13. Iritani N, Yamamoto SP, Abe N, et al. GII.17 norovirus infections in outbreaks of acute nonbacterial gastroenteritis in Osaka City, Japan during two decades. J Med Virol. 2019;91(12):2101-2107. doi: 10.1002/jmv.25560
14. Kim H, Won YJ, Kang LH, et al. Complete sequence analysis of human norovirus GII.17 detected in South Korea. Epidemiol Infect. 2019;147:e203. doi: 10.1017/S0950268819000943
15. LeBlanc JJ, Pettipas J, Gaston D, et al. Outbreak of norovirus GII.P17-GII.17 in the Canadian Province of Nova Scotia. Can J Infect Dis Med Microbiol. 2016;2016:1280247. doi: 10.1155/2016/1280247
16. Giammanco GM, De Grazia S, Bonura F, et al. Norovirus GII.17 as major epidemic strain in Italy, winter 2015–16. Emerg Infect Dis. 2017;23(7):1206-1208. doi: 10.3201/eid2307.161255
17. Makhaola K, Moyo S, Lechiile K, Goldfarb DM, Kebaabetswe LP. Genetic and epidemiological analysis of norovirus from children with gastroenteritis in Botswana, 2013–2015. BMC Infect Dis. 2018;18(1):246. doi: 10.1186/s12879-018-3157-y
18. Gelaw A, Pietsch C, Mann P, Liebert UG. Molecular detection and characterisation of sapoviruses and noroviruses in outpatient children with diarrhoea in Northwest Ethiopia. Epidemiol Infect. 2019;147:e218. doi: 10.1017/S0950268819001031
19. Lee S, Jang J, Bae K, Lee W, Chung H, Park S. Prevalence of human Norovirus by genotype in contaminated groundwater in Korea over the last decade (2007–2016). J Microbiol. 2018;56(12):926-931. doi: 10.1007/s12275-018-8340-8
20. Mabasa VV, Meno KD, Taylor MB, Mans J. Environmental surveillance for noroviruses in selected South African wastewaters 2015–2016: Emergence of the novel GII.17. Food Environ Virol. 2018;10(1):16-28. doi: 10.1007/s12560-017-9316-2
21. Hasing ME, Lee BE, Qiu Y, et al. Changes in norovirus genotype diversity in gastroenteritis outbreaks in Alberta, Canada: 2012–2018. BMC Infect Dis. 2019;19(1):177. doi: 10.1186/s12879-019-3792-y
22. Бутакова Л.В., Сапега Е.Ю., Троценко О.Е., Зайцева Т.А., Каравянская Т.Н., Лебедева Л.А. Водная вспышка острой кишечной инфекции, обусловленная рекомбинантным норовирусом генотипа GII.P7-GII.6, в городе Хабаровске в 2019 году // Здоровье населения и среда обитания. 2020. № 6 (327). С. 50–54. doi: 10.35627/2219-5238/2020-327-6-50-54
23. Fumian TM, Ferreira FC, de Andrade JDSR, et al. Norovirus foodborne outbreak associated with the consumption of ice pop, Southern Brazil, 2020. Food Environ Virol. 2021;13(4):553-559. doi: 10.1007/s12560-021-09495-9
24. Zhou X, Kong DG, Li J, et al. An outbreak of gastroenteritis associated with GII.17 norovirus-contaminated secondary water supply system in Wuhan, China, 2017. Food Environ Virol. 2019;11(2):126-137. doi: 10.1007/s12560-019-09371-7
25. Sang S, Yang X. Evolutionary dynamics of GII.17 norovirus. PeerJ. 2018;6:e4333. doi: 10.7717/peerj.4333
26. Chen C, Wu B, Zhang H, et al. Molecular evolution of GII.P17-GII.17 norovirus associated with sporadic acute gastroenteritis cases during 2013–2018 in Zhoushan Islands, China. Virus Genes. 2020;56(3):279-287. doi: 10.1007/s11262-020-01744-6
Об авторах
А. С. ВодопьяновРоссия
Водопьянов Алексей Сергеевич – к.м.н., ведущий научный сотрудник лаборатории молекулярной биологии
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002, Российская Федерация
Р. В. Писанов
Россия
Писанов Руслан Вячеславович – к.м.н., заведующий лабораторией молекулярной биологии
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002, Российская Федерация
С. О. Водопьянов
Россия
Водопьянов Сергей Олегович – д.м.н., главный научный сотрудник лаборатории микробиологии холеры
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002, Российская Федерация
О. С. Чемисова
Россия
Чемисова Ольга Сергеевна – к.б.н., заведующий лабораторией «Коллекция патогенных микроорганизмов»
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002, Российская Федерация
А. А. Герасименко
Россия
Герасименко Артем Александрович – младший научный сотрудник лаборатории
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002, Российская Федерация
А. К. Носков
Россия
Носков Алексей Кимович – к.м.н., директор
ул. М. Горького, д. 117/40, г. Ростов-на-Дону, 344002, Российская Федерация
С. С. Слись
Россия
Слись Сергей Сергеевич – главный специалист-эксперт отдела эпидемиологического надзора
ул. 18-я линия, д. 17, г. Ростов-на-Дону, 344019, Российская Федерация
С. А. Ненадская
Россия
Ненадская Светлана Алексеевна – начальник отдела эпидемиологического надзора
ул. 18-я линия, д. 17, г. Ростов-на-Дону, 344019, Российская Федерация
А. Д. Коренева
Россия
Коренева Анастасия Дмитриевна – главный специалист-эксперт отдела эпидемиологического надзора
ул. 18-я линия, д. 17, г. Ростов-на-Дону, 344019, Российская Федерация
А. В. Коломойцева
Россия
Коломойцева Алина Владимировна – ведущий специалист-эксперт отдела эпидемиологического надзора
ул. 18-я линия, д. 17, г. Ростов-на-Дону, 344019, Российская Федерация
Е. В. Ковалев
Россия
Ковалев Евгений Владимирович – руководитель
ул. 18-я линия, д. 17, г. Ростов-на-Дону, 344019, Российская Федерация
А. Р. Литовко
Россия
Литовко Анна Радиковна – заведующий лабораторией вирусологических исследований
ул. 7-я линия, д. 67, г. Ростов-на-Дону, 344019, Российская Федерация
Н. В. Половинка
Россия
Половинка Нина Владимировна – заведующий отделом эпидемиологии и экспертизы
ул. 7-я линия, д. 67, г. Ростов-на-Дону, 344019, Российская Федерация
Рецензия
Для цитирования:
Водопьянов А.С., Писанов Р.В., Водопьянов С.О., Чемисова О.С., Герасименко А.А., Носков А.К., Слись С.С., Ненадская С.А., Коренева А.Д., Коломойцева А.В., Ковалев Е.В., Литовко А.Р., Половинка Н.В. Оценка генетического разнообразия возбудителя норовирусной инфекции в пунктах временного размещения беженцев в Ростовской области в 2022 году с помощью онлайн-программы NoroNetRus. Здоровье населения и среда обитания – ЗНиСО. 2022;1(11):82-88. https://doi.org/10.35627/2219-5238/2022-30-11-82-88
For citation:
Vodop’ianov A.S., Pisanov R.V., Vodop’ianov S.O., Chemisova O.S., Gerasimenko A.A., Noskov A.K., Slis S.S., Nenadskaya S.A., Koreneva A.D., Kolomoitseva A.V., Kovalev E.V., Litovko A.R., Polovinka N.V. Assessment of Genetic Diversity of Noroviruses Circulating in Temporary Accommodation Centers for Refugees in the Rostov Region in 2022 Using the NoroNetRus Online Software. Public Health and Life Environment – PH&LE. 2022;1(11):82-88. (In Russ.) https://doi.org/10.35627/2219-5238/2022-30-11-82-88









.png)



























