Перейти к:
Генетическое профилирование штаммов Salmonella enteritidis, выделенных на территории Ставропольского края в 2016–2019 гг.
https://doi.org/10.35627/2219-5238/2022-30-6-66-71
Аннотация
Введение. В Ставропольском крае наиболее частым этиологическим агентом сальмонеллезов у людей является S. enteritidis, что соответствует общемировой тенденции. В связи с этим определение серовара дает мало информации в ходе эпидемиологического расследования и обуславливает необходимость субвидового типирования изолятов S. enteritidis.
Цель исследования: провести MLVA-типирование штаммов S. enteritidis, выделенных на территории Ставропольского края в 2016–2019 гг., проанализировать генетическую структуру популяции сальмонелл в регионе.
Материалы и методы. Исследовано 122 штамма S. enteritidis, изолированных в 2016–2019 гг. из проб испражнений больных острыми кишечными инфекциями в Ставропольском крае (г. Ставрополь и регион Кавказских Минеральных Вод (КМВ)). MLVA-типирование осуществляли по 5 вариабельным локусам. Размер амплифицированных локусов определяли методом капиллярного электрофореза.
Результаты. Исследуемые штаммы отличались высокой генетической гетерогенностью и относились к 25 MLVA-генотипам. На территории г. Ставрополя выявлены штаммы S. enteritidis принадлежащие к 24 MLVA-типам. Доминирующим геновариантом в г. Ставрополе являлся 3-10-5-4-1, к которому относилось 40 штаммов сальмонелл (44,4 %), выделенных в 2016–2019 гг. В отдельные годы возрастал удельный вес других геновариантов. В регионе КМВ были выделены штаммы, относящиеся к 7 MLVA-генотипам, большинство выделенных культур принадлежало к генотипу 3-10-5-4-1. В регионе КМВ ежегодно отмечалась смена доминирующего варианта S. enteritidis. Доминирующие на территории края MLVA-типы широко распространены в мире и обладают значительным эпидемическим потенциалом.
Заключение. Получены новые данные о MLVA-генотипах S. enteritidis, встречающихся на территории Ставропольского края (г. Ставрополь и регион КМВ). Определены доминирующие в регионе геноварианты, отмечены различия в соотношении циркулирующих MLVA-генотипов S. enteritidis в 2016–2019 гг., это определяет необходимость молекулярно-генетического мониторинга для оценки динамических изменений генетической структуры популяции сальмонелл в режиме реального времени. Полученные в результате работы данные могут быть использованы при эпидемиологическом анализе возможных случаев (вспышек) сальмонеллеза для определения источника и путей распространения инфекции.
Ключевые слова
Для цитирования:
Чекрыгина Е.В., Васильева О.В., Волынкина А.С., Алехина Ю.А., Куличенко А.Н. Генетическое профилирование штаммов Salmonella enteritidis, выделенных на территории Ставропольского края в 2016–2019 гг. Здоровье населения и среда обитания – ЗНиСО. 2022;(6):66-71. https://doi.org/10.35627/2219-5238/2022-30-6-66-71
For citation:
Chekrygina E.V., Vasilyeva O.V., Volynkina A.S., Alekhina Yu.A., Kulichenko A.N. Genetic Profiling of Salmonella enteritidis Strains Isolated in the Stavropol Region in 2016–2019. Public Health and Life Environment – PH&LE. 2022;(6):66-71. (In Russ.) https://doi.org/10.35627/2219-5238/2022-30-6-66-71
Введение. Занимающий одно из ведущих мест в структуре острых кишечных бактериальных инфекций сальмонеллез характеризуется сложностью этиологической структуры, полиморфизмом клинических проявлений и формированием бактерионосительства. В России, несмотря на тенденцию к снижению распространения, сальмонеллезы занимают третье место (после ротаи норовирусной инфекции) в структуре заболеваемости с фекально-оральным механизмом передачи возбудителя1.
Для Ставропольского края сальмонеллез остается значимой инфекционной патологией с высокими эпидемиологическими рисками формирования очагов с множественными случаями заболеваний. В 2019 году произошел двукратный рост заболеваемости по сравнению с 2016–2018 гг.
На территории Ставропольского края находятся Кавказские Минеральные Воды (КМВ) – группа курортов федерального значения, особо охраняемый эколого-курортный регион Российской Федерации. Интенсивное развитие туристической отрасли края существенно увеличивает угрозу возникновения эпидемических ситуаций в регионе, в т. ч. массовых вспышек ОКИ, и обуславливает необходимость совершенствования системы эпидемиологического надзора и внедрения эффективного алгоритма идентификации штаммов для расследования вспышек и отдельных случаев заболевания ОКИ.
В Ставропольском крае наиболее частым этиологическим агентом сальмонеллезов у людей является Salmonella enterica subsp. enterica serovar enteritidis (S. enteritidis), что соответствует общемировой тенденции [1]. Этот серовар выделяется в 84,1 % всех диагностированных случаев сальмонеллеза в крае2. В связи с этим определение серовара дает мало информации в ходе эпидемиологического расследования и обуславливает необходимость субвидового типирования изолятов S. enteritidis.
В последние годы широкое практическое применение для субвидовой характеристики микроорганизмов получили методы молекулярно-генетического анализа, обладающие большей дифференцирующей способностью по сравнению с фенотипическими методами исследования.
Молекулярное типирование S. enteritidis наиболее часто проводят с использованием методов гель-электрофореза в пульсирующем поле (PFGE-типирование), мультилокусного анализа числа вариабельных тандемных повторов (Multi Locus Variable Number Tandem Repeat Analysis – MLVA) и полногеномного секвенирования (Whole Genome Sequencing – WGS) [2][3]. Применяемый для определения подтипов сальмонелл метод PFGE трудоемок, требует наличия специализированного оборудования и обученного персонала, время выполнения анализа составляет около трех суток [4]. Метод WGS обладает наибольшей дискриминирующей способностью, однако является дорогостоящим, требует проведения сложного биоинформационного анализа и пока не подходит для рутинного субвидового типирования S. enteritidis [5].
MLVA-типирование основано на анализе локусов с вариабельным числом тандемных повторов (VNTR-variable number tandem repeats). VNTR-локусы являются важным маркером полиморфизма генов, используемых для типирования близкородственных штаммов. Метод MLVA является основным для типирования сальмонелл в странах Европы, что связано с быстротой выполнения анализа и высокой воспроизводимостью результатов [6][7]. Анализ MLVA-профилей штаммов позволяет идентифицировать клоны и дифференцировать штаммы при расследовании вспышек, эффективно устанавливать или исключать связь между отдельными случаями заболеваний [8][9][10]. В странах ЕС разработаны стандартные операционные процедуры по выполнению MLVA-типирования сероваров S. enteritidis и S. typhimurium [11].
Для эффективного применения молекулярно-генетических методов при решении различных эпидемиологических задач необходимо накопление сведений об особенностях генотипов штаммов, циркулирующих в исследуемом регионе, а также информации о генетической гетерогенности возбудителя в мире. Молекулярное типирование штаммов S. enteritidis, выделенных на территории Ставропольского края, позволит получить новую информацию о генетической структуре популяции сальмонелл в регионе, сформировать базу данных для молекулярного анализа при эпидемиологическом расследовании случаев заболевания ОКИ.
Цель исследования: провести MLVA-типирование штаммов S. enteritidis, выделенных на территории Ставропольского края в 2016–2019 гг., проанализировать генетическую структуру популяции сальмонелл в регионе.
Материалы и методы.
В работе использованы 122 штамма S. enteritidis, изолированных в 2016–2019 гг. из проб испражнений больных острыми кишечными инфекциями в Ставропольском крае, в т. ч. в г. Ставрополе (90 штаммов) и регионе КМВ (32 штамма). Случаи ОКИ эпидемиологически не связаны между собой. В этот период отмечалась спорадическая заболеваемость ОКИ, очаги групповой заболеваемости ОКИ не зарегистрированы.
Видовую идентификацию проводили по ферментативной активности в отношении различных субстратов с использованием наборов реагентов для идентификации энтеробактерий ММТЕ1 и ММТЕ2 (НПО «Аллерген», г. Ставрополь). Определение антигенной структуры штаммов выполняли в реакции агглютинации с сыворотками диагностическими сальмонеллезными адсорбированными к О- и Н-антигенам сальмонелл «ПЕТСАЛ» (производства СПбНИИВС Россия).
Выделение бактериальной ДНК производили с помощью набора реагентов «ДНК-Сорб-В» (производства ФБУН «ЦНИИЭ» Роспотребнадзора, г. Москва, Россия). Полученные образцы ДНК использовали для амплификации фрагментов генома изучаемых штаммов S. enteritidis.
MLVA-типирование проводили в соответствии с протоколом, разработанным Hopkins K.L. [12]. Для амплификации VNTR-локусов использовали праймеры, указанные в статье Malorny B. [13] (табл. 1). Размер амплифицированных локусов определяли методом капиллярного электрофореза на секвенаторе Applied Biosystems 3500 (США). Точный размер различающихся по длине аллелей определяли секвенированием по Сэнгеру с использованием Big Dye Terminator v 3.1 Cycle Sequencing Kit (Applied Biosystems). Индивидуальный MLVA-генотип штамма был получен на основании числа тандемных повторов в локусах: SENTR7-SENTR5-SENTR6-SENTR4- SE3.
Таблица 1. Последовательности праймеров для амплификации VNTR-локусов
Table 1. Primer sequences for amplification of VNTR loci

Результаты. Проведено MLVA-типирование 122 штаммов S. enteritidis, выделенных от больных ОКИ в Ставропольском крае с 2016 по 2019 г. Исследованные штаммы отличались высокой генетической гетерогенностью и относились к 25 MLVA-генотипам. Наибольшее количество разных аллельных вариантов (8) выявлено в локусе SENTR5. В локусе SENTR6 выявлено 6 разных аллелей, в SENTR4 – 4, в локусах SENTR7 и SE3 – по 2.
На территории г. Ставрополя выявлены штаммы S. enteritidis, принадлежащие к 24 MLVA-типам, 74,4 % штаммов, выделенных в г. Ставрополе за весь период наблюдений, относились к семи наиболее распространенным MLVA-генотипам (табл. 2). Также выявлены минорные геноварианты, доля которых составляла 1,1–2,2 % от общего количества исследованных изолятов (в совокупности 25,6 %).
Доминирующим геновариантом в г. Ставрополе являлся 3-10-5-4-1, к которому относилось 40 штаммов сальмонелл (44,4 %), выделенных в 2016–2019 гг. В отдельные годы возрастал удельный вес других геновариантов: 2-10-8-3-2, 3-10-5-3-1, 3-11-5-4-1, 3-10-5-3-1, 2-10-14-6-1, 2-12-8-3-2 и 3-18-5-4-1. Ежегодно с 2016 по 2019 г. на территории г. Ставрополя встречались штаммы S. enteritidis, принадлежащие к MLVAгенотипам 3-10-5-4-1 и 3-9-5-4-1. В 2016 г. выявлены 11 геновариантов сальмонелл, преобладали штаммы с MLVA-генотипами 3-10-5-4-1 (36,4 %) и 2-10- 8-3-2 (13,6 %). В 2017 г. исследованные культуры принадлежали к 14 геновариантам, доминирующими являлись MLVA-типы 3-10-5-4-1 (53,5 %) и 3-10-5-3-1 (11,6 %). В 2018 г. выявлено 7 геновариантов S. enteritidis, отмечались изменения в соотношении генетических типов сальмонелл, доминирующим вариантом являлся 2-10-8-3-2 (23,1 %), доля варианта 3-10-5-4-1 снизилась до 15,4 %, выявлены штаммы MLVA-типов 2-12-8- 3-2 и 3-18-5-4-1 (по 2 штамма, 15,4 %). В 2019 г. исследованные штаммы относились к 4 вариантам, преобладал 3-10-5-4-1 (58,3 %), доля варианта 3-11-5-4-1 возросла до 25,0 %.
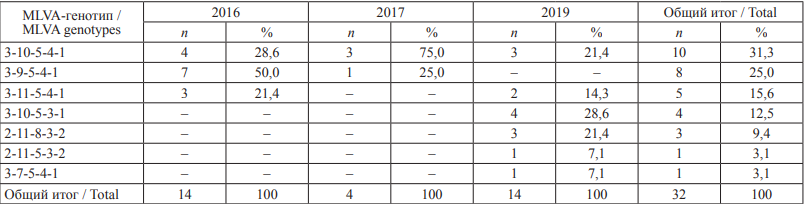
В регионе КМВ в 2016–2019 гг. выявлены штаммы S. enteritidis, принадлежащие к 7 MLVA-типам, большинство культур относилось к МLVA-генотипу 3-10-5-4-1 (31,3 %). В 2016 г. в городах-курортах КМВ выделены культуры S. enteritidis, относящиеся к трем MLVA-типам, преобладали штаммы геноварианта 3-9-5-4-1 (50,0 %). В 2017 г. доминирующим вариантом являлся 3-10-5-4-1 (75,0 %). В 2019 г. выявлено шесть геновариантов сальмонелл, преобладали штаммы генотипа 3-10-5-3-1 (28,6 %) (табл. 3).
Таблица 2. MLVA-генотипы S. enteritidis, выявленные в г. Ставрополе в 2016–2019 гг.
Table 2. MLVA genotype of S. enteritidis isolated in Stavropol in 2016–2019
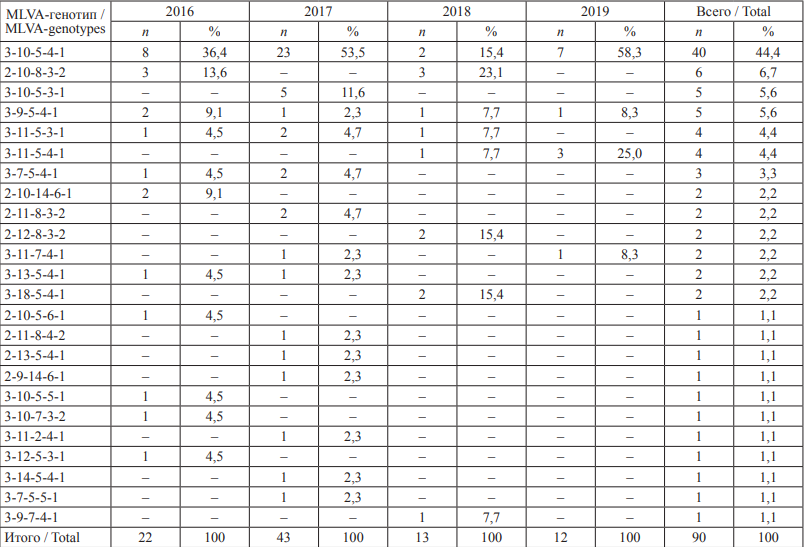
Таблица 3. MLVA-генотипы S. enteritidis, выявленные в регионе КМВ в 2016–2019 гг.
Table 3. MLVA genotypes of S. enteritidis isolated in the Caucasian Mineral Waters in 2016–2019
Обсуждение. Метод MLVA-типирования широко применяется для оценки генетической гетерогенности штаммов S. enteritidis в мире, в том числе для выявления эпидемических геновариантов сальмонелл, способных вызывать вспышки сальмонеллеза на территории нескольких государств, и отслеживания возникновения новых генетических вариантов. Для накопления, хранения и обмена результатами MLVA-типирования сальмонелл, выделенных в странах ЕС и других регионах, а также для принятия управленческих решений в области общественного здравоохранения был разработан модуль Enter-net на платформе The European Surveillance System (TESSy) [14].
Ретроспективное генетическое типирование S. enteritidis в Европе позволило выявить эпидемические варианты сальмонелл, преобладающие на территории большинства европейских государств, а также многочисленные минорные варианты, вызывающие спорадические случаи заболевания сальмонеллезами. Так, штаммы S. enteritidis, относящиеся к MLVA-типам 3-10-5-4- 1, 3-11-5-4-1, 2-10-7-3-2, 3-9-5-4-1, доминировали в Португалии в период с 2012 по 2019 г. [15], являлись наиболее часто выявляемыми вариантами в Бельгии в 2007–2012 гг. [16], в Венгрии и Австрии в 2016–2017 гг.
В 2010 г. в ЕС были выявлены штаммы S. enteritidis, относящиеся к новым MLVA-типам: 2-10-6-3-2, 2-10-8-3-2 и 2-11-8-3-2, вызвавшие в 2016–2017 гг. вспышку сальмонеллеза, охватившую территорию большинства европейских стран [17].
Отмечена корреляция между увеличением доли эпидемических MLVA-типов S. enteritidis в регионе и ростом числа случаев сальмонеллезов [10].
Встречающиеся на территории края MLVAтипы (3-10-5-4-1, 3-9-5-4-1, 3-11-5-4-1 и 2-10-8- 3-2) широко распространены в мире, обладают значительным эпидемическим потенциалом. Менее распространенный геновариант 2-11-8-3-2, доля которого увеличилась в г. Ставрополе в 2018 г., вызвал рост заболеваемости S. enteritidis PT-8 в странах ЕС в 2017 г. [18].
В Российской Федерации для проведения молекулярно-генетического мониторинга за циркуляцией штаммов S. enteritidis используются методы PFGE и MLVA [19][20]. В 2011–2019 гг. при поведении субтипирования штаммов методом PFGE c помощью эндонуклеаз рестрикции XbaI BlnI в РФ выделены S. enteritidis, относящиеся к 102 PFGE-типам. Наиболее распространенным у человека являлся субтип JEG01/001/JECA26/001, доля которого составляла 44 % от всех клинических изолятов S. enteritidis в РФ. Отмечались существенные различия в разнообразии выделяемых PFGE-типов в отдельных регионах страны [21].
В результате работы получены новые данные о MLVA-генотипах S. enteritidis, встречающихся на территории Ставропольского края (г. Ставрополь и регион КМВ). Большинство клинических изолятов S. enteritidis, выделенных в 2016–2019 гг. на территории г. Ставрополя и региона КМВ, принадлежали к MLVA-генотипу 3-10-5-4-1, их доля в г. Ставрополе составила 44,4 %, в регионе КМВ – 31,3 %. На территории г. Ставрополя отмечено доминирование данного варианта в 2016, 2017, 2019 гг., и только в 2018 г. превалирующим вариантом стал 2-10-8-3-2. В регионе КМВ ежегодно отмечалась смена доминирующего варианта, что может быть связано с высокой миграционной активностью населения и относительно небольшим количеством исследованных штаммов.
Ретроспективное исследованиe штаммов S. enteritidis, выделенных на территории Ставропольского края в 2016–2019 гг., показало, что большинство изученных штаммов сальмонелл (100 штаммов, 81,96 %) относятся к восьми наиболее распространенным генотипам. Количественное соотношение MLVA-типов S. enteritidis, изолированных на территории Ставропольского края, в целом схоже с соотношением PFGE-генотипов S. enteritidis, выявленных в РФ: к одному преобладающему MLVA-типу относятся 41 % штаммов, изолированных в Ставропольском крае. В России в последние годы также установлено превалирование одного PFGE-субтипа S. enteritidis [21].
Регулярный мониторинг за возникновением новых геновариантов сальмонелл, способных обладать повышенной вирулентностью и быть причиной изменений в патогенезе заболевания, является неотъемлемой частью современного эпиднадзора за ОКИ. Он имеет большое значение для здравоохранения в плане прогнозирования возможных сценариев развития эпидситуации с целью профилактики заболеваемости ОКИ бактериальной этиологии.
Метод MLVA показал высокую разрешающую способность при субтипировании штаммов S. enteritidis [19][20]. В то же время наличие космополитных штаммов может затруднять расследование вспышек, и в некоторых случаях необходимо выполнение WGS (whole genome sequencing).
Заключение. Таким образом, в результате проведенных исследований получены новые данные о MLVA-генотипах S. enteritidis, встречающихся на территории Ставропольского края (г. Ставрополь и регион КМВ). Определены доминирующие в регионе геноварианты, отмечены различия в соотношении циркулирующих MLVA-генотипов S. enteritidis в 2016–2019 гг. Это диктует необходимость постоянного молекулярно-генетического мониторинга для оценки динамических изменений генетической структуры популяции сальмонелл в режиме реального времени.
Применение метода MLVA-типирования штаммов позволит не только проводить анализ генетической структуры популяции сальмонелл, но и эффективно устанавливать или исключать связь между отдельными случаями заболеваний, прогнозировать развитие эпидемиологической ситуации на основании данных о соотношении геновариантов сальмонелл в регионе. Полученные в результате работы данные могут быть использованы при эпидемиологическом анализе возможных случаев (вспышек) сальмонеллеза для определения источника и путей распространения инфекции.
1. О состоянии санитарно-эпидемиологического благополучия населения в Российской Федерации в 2019 году: государственный доклад. М.: Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека, 2020. 299 с. Доступно по: https://www.rospotrebnadzor.ru/upload/iblock/8e4/gosdoklad-za-2019_seb_ 29_05.pdf43 (дата доступа: 20.04.2022).
2. О состоянии санитарно-эпидемиологического благополучия населения в Ставропольском крае в 2019 году: государственный доклад. Ставрополь, 2019. 107 с. Доступно по: http://26.rospotrebnadzor.ru/doc/gd/Gosdoklad_2019.pdf (дата доступа: 20.04.2022).
Список литературы
1. Hendriksen RS, Vieira AR, Karlsmose S, et al. Global monitoring of Salmonella serovar distribution from the World Health Organization Global Foodborne Infections Network Country Data Bank: results of quality assured laboratories from 2001 to 2007. Foodborne Pathog Dis. 2011;8(8):887-900. doi: 10.1089/fpd.2010.0787
2. Ziebell K, Chui L, King R, Johnson S, Boerlin P, Johnson RP. Subtyping of Canadian isolates of Salmonella Enteritidis using Multiple Locus Variable Number Tandem Repeat Analysis (MLVA) alone and in combination with Pulsed-Field Gel Electrophoresis (PFGE) and phage typing. J Microbiol Methods. 2017;139:29-36. doi: 10.1016/j.mimet.2017.04.012
3. Inns T, Ashton PM, Herrera-Leon S, et al. Prospective use of whole genome sequencing (WGS) detected a multi-country outbreak of Salmonella Enteritidis. Epidemiol Infect. 2017;145(2):289-298. doi: 10.1017/S0950268816001941
4. Wattiau P, Boland C, Bertrand S. Methodologies for Salmonella enterica subsp. enterica subtyping: gold standards and alternatives. Appl Environ Microbiol. 2011;77(22):7877-7885. doi: 10.1128/AEM.05527-11
5. den Bakker HC, Allard MW, Bopp D, et al. Rapid whole-genome sequencing for surveillance of Salmonella Enterica serovar enteritidis. Emerg Infect Dis. 2014;20(8):1306-1314. doi: 10.3201/eid2008.131399
6. Ferrari RG, Panzenhagen PHN, Conte-Junior CA. Phenotypic and genotypic eligible methods for Salmonella Typhimurium source tracking. Front Microbiol. 2017;8:2587. doi: 10.3389/fmicb.2017.02587
7. Lienemann T, Kyyhkynen A, Halkilahti J, Haukka K, Siitonen A. Characterization of Salmonella Typhimurium isolates from domestically acquired infections in Finland by phage typing, antimicrobial susceptibility testing, PFGE and MLVA. BMC Microbiol. 2015;15:131. doi: 10.1186/s12866-015-0467-8
8. Liu Y, Shi X, Li Y, et al. The evaluation and application of multilocus variable number tandem repeat analysis (MLVA) for the molecular epidemiological study of Salmonella enterica subsp. enterica serovar Enteritidis infection. Ann Clin Microbiol Antimicrob. 2016;15:4. doi: 10.1186/s12941-016-0119-3
9. Mughini-Gras L, Franz E, van Pelt W. New paradigms for Salmonella source attribution based on microbial subtyping. Food Microbiol. 2018;71:60-67. doi: 10.1016/j.fm.2017.03.002
10. Muvhali M, Smith AM, Rakgantso AM, Keddy KH. Investigation of Salmonella Enteritidis outbreaks in South Africa using multi-locus variable-number tandem-repeats analysis, 2013–2015. BMC Infect Dis. 2017;17(1):661. doi: 10.1186/s12879-017-2751-8
11. European Centre for Disease Prevention and Control. ECDC Technical Document. Laboratory standard operating procedure for multiple-locus variable-number tandem repeat analysis of Salmonella enterica serotype Enteritidis. Stockholm: ECDC; 2016. doi: 10.2900/973540
12. Hopkins KL, Peters TM, de Pinna E, Wain J. Standardisation of multilocus variable-number tandem-repeat analysis (MLVA) for subtyping of Salmonella enterica serovar Enteritidis. Euro Surveill. 2011;16(32):19942.
13. Malorny B, Junker E, Helmuth R. Multi-locus variable-number tandem repeat analysis for outbreak studies of Salmonella enterica serotype Enteritidis. BMC Microbiol. 2008;8:84. doi: 10.1186/1471-2180-8-84
14. Peters T, Bertrand S, Björkman JT, et al. Multi-laboratory validation study of multilocus variable-number tandem repeat analysis (MLVA) for Salmonella enterica serovar Enteritidis, 2015. Euro Surveill. 2017;22(9):30477. doi: 10.2807/1560-7917.ES.2017.22.9.30477
15. Silveira LS. Phenotypic and genotypic characterization of Salmonella spp. isolates in Portugal. Doctor of Biology thesis. NOVA University Lisbon; 2019. Accessed April, 25, 2022. http://hdl.handle.net/10362/91582
16. Bertrand S, De Lamine de Bex G, Wildemauwe C, et al. Multi locus variable-number tandem repeat (MLVA) typing tools improved the surveillance of Salmonella enteritidis: a 6 years retrospective study. PLoS One. 2015;10(2):e0117950. doi: 10.1371/journal.pone.0117950
17. European Food Safety Authority, European Centre for Disease Prevention and Control Multi-country outbreak of Salmonella Enteritidis phage type 8, MLVA profile 2-9-7-3-2 and 2-9-6-3-2 infections. Technical report EFSA. EFSA Supporting Publications. 2017;14(3):1188E. doi: 10.2903/sp.efsa.2017.EN-1188
18. Usein CR, Oprea M, Ciontea AS, et al. A snapshot of the genetic diversity of Salmonella Enteritidis population involved in human infections in Romania taken in the European epidemiological context. Pathogens. 2021;10(11):1490. doi: 10.3390/pathogens10111490
19. Кулешов К.В., Браславская С.И., Подколзин А.Т. Разработка и апробация методики MLVA-анализа с целью субтипирования изолятов S. Enteritidis. Молекулярная диагностика – 2010, Москва, 24–26 ноября 2010 года / Под редакцией В.И. Покровского. Москва: Киселева Н.В. 2010. С. 350–353.
20. Кулешов К.В., Подколзин А.Т., Рожнова. С.Ш. Сравнительная оценка молекулярно-генетических методов типирования изолятов S. Enteritidis в очагах групповой заболеваемости. Молекулярная диагностика – 2010, Москва, 24–26 ноября 2010 года / Под редакцией В.И. Покровского. Москва: Киселева Н.В. 2010. С. 353–357.
21. Рожнова С.Ш., Кулешов К.В., Павлова А.С. и др. Гетерогенность изолятов нетифоидных сальмонелл из различных источников выделения в Российской Федерации в 2010–2019 гг. // Эпидемиология и инфекционные болезни. 2020. Т. 25. № 1. С. 26–34. doi: 10.17816/EID35184
Об авторах
Е. В. ЧекрыгинаРоссия
Чекрыгина Елена Владимировна – ассистент кафедры микробиологии
ул. Мира, д. 310, г. Ставрополь, 355017
О. В. Васильева
Россия
Васильева Оксана Васильевнa – к.м.н., заведующая лаборатории диагностики бактериальных инфекций
ул. Советская, д. 13–15, г. Ставрополь, 355035
А. С. Волынкина
Россия
Волынкина Анна Сергеевна – к.б.н., заведующая лабораторией диагностики вирусных инфекций
ул. Советская, д. 13–15, г. Ставрополь, 355035
Ю. А. Алехина
Россия
Алехина Юлия Александровна – врач КЛД лаборатории диагностики бактериальных инфекций
ул. Советская, д. 13–15, г. Ставрополь, 355035
А. Н. Куличенко
Россия
Куличенко Александр Николаевич – д.м.н., профессор, директор
ул. Советская, д. 13–15, г. Ставрополь, 355035
Рецензия
Для цитирования:
Чекрыгина Е.В., Васильева О.В., Волынкина А.С., Алехина Ю.А., Куличенко А.Н. Генетическое профилирование штаммов Salmonella enteritidis, выделенных на территории Ставропольского края в 2016–2019 гг. Здоровье населения и среда обитания – ЗНиСО. 2022;(6):66-71. https://doi.org/10.35627/2219-5238/2022-30-6-66-71
For citation:
Chekrygina E.V., Vasilyeva O.V., Volynkina A.S., Alekhina Yu.A., Kulichenko A.N. Genetic Profiling of Salmonella enteritidis Strains Isolated in the Stavropol Region in 2016–2019. Public Health and Life Environment – PH&LE. 2022;(6):66-71. (In Russ.) https://doi.org/10.35627/2219-5238/2022-30-6-66-71









.png)



























